nnUNet首先进行plan_and_preprocess来对数据进行预处理,生成预处理(裁剪,分析数据属性)后的中间阶段数据。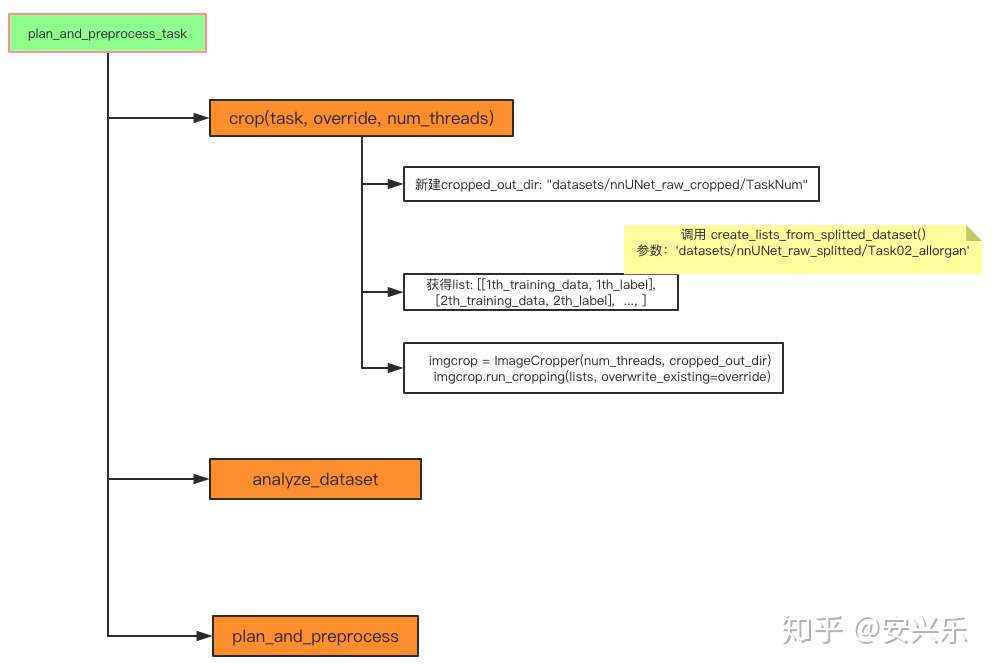
# task: Task02_allorgan
crop(task, override=override, num_threads=processes_lowres)
analyze_dataset(task, override, collect_intensityproperties=True, num_processes=processes_lowres)
plan_and_preprocess(task, processes_lowres, processes_fullres, no_preprocessing)
我们接下来从analyze_dataset()函数开始,
# plan_and_preprocess_task.py
def analyze_dataset(task_string, override=False, collect_intensityproperties=True, num_processes=8):
cropped_out_dir = join(cropped_output_dir, task_string)
dataset_analyzer = DatasetAnalyzer(cropped_out_dir, overwrite=override, num_processes=num_processes)
_ = dataset_analyzer.analyze_dataset(collect_intensityproperties)
analyze_dataset函数调用外部的DatasetAnalyzer.py.DatasetAnalyzer()类来实现。该类主要分析训练数据集的数据统计分布特征,像体素均值、中值、最大值、最小值、0.95/0.05分位值、类别数、缩放比例等。
DatasetAnalyzer类
__init__() 初始化不必多说,设置分析的进程数,裁切后的数据位置,数据前缀识别符(case_xxx_0000.nii.gz去掉_0000.nii.gz)。
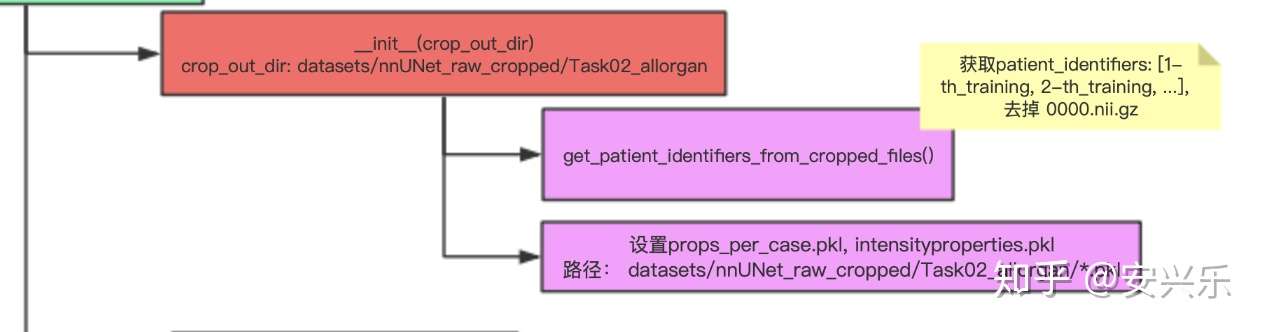
之后,主程序调用analyze_dataset(),开始对数据进行分析,将结果保存入nnUNet_raw_cropped/dataset_properties.pkl。放图:
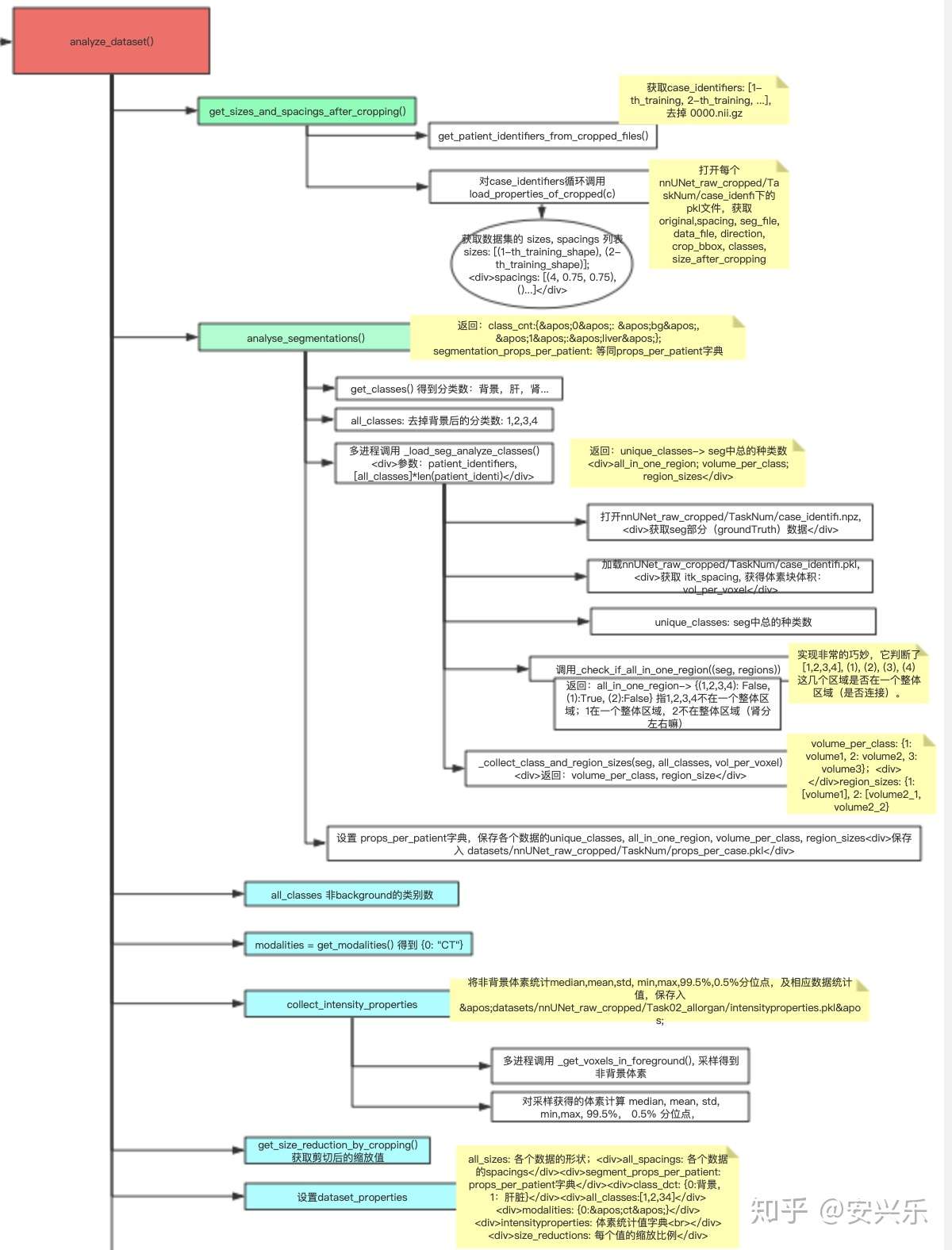
错略看一下,该函数大约调用了get_size_and_spacings_after_cropping(),analyse_segmentations(),get_modalities(),collect_intensity_properties(),get_size_reduction_by_cropping()。
我们首先进入get_sizes_and_spacings_after_cropping(),该函数主要对裁切后数据的sizes,spacings进行统计。特别注意,我们接下来将层层递进,不断“入栈出栈”来逐层分析各个函数。
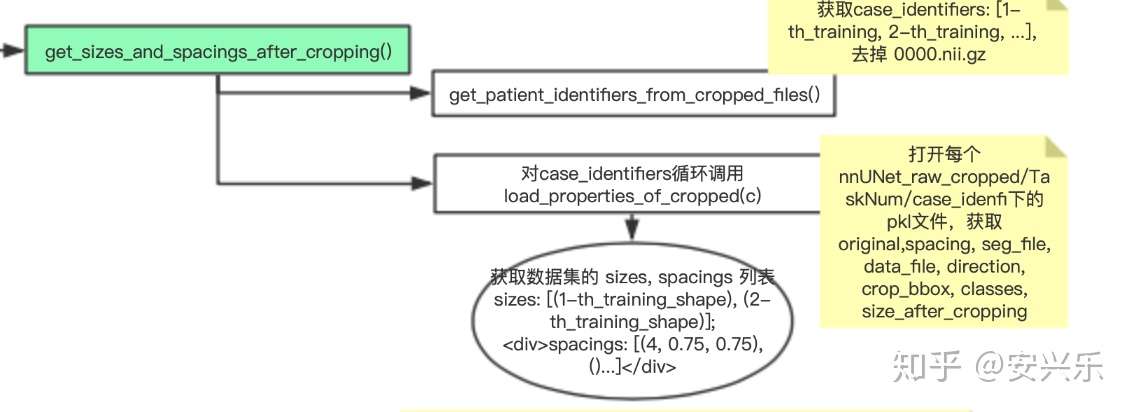
这里第二次调用 get_patient_identifiers_from_cropped_files(),实际上在类初始化时就已经调用了一次了,而且参数一样!这里很明显冗余了,完全没有必要调用,直接把self.patient_identifiers拿过来就行了!已提交pull requests请求,希望作者能去掉。然后对每个case_identifiers循环调用load_properties_of_cropped(c),打开nnUNet_raw_cropped下的Task02_allorgan获取数据集的pkl文件,调用self.load_properties_of_cropped()来获取数据的itk_origin、itk_spacing、itk_direction、crop_bbox等属性,这里将其添加入 sizes,spacings列表并返回。
终于进入重头戏analyse_segmentations(),这里对mask进行详细分析。我们打开代码: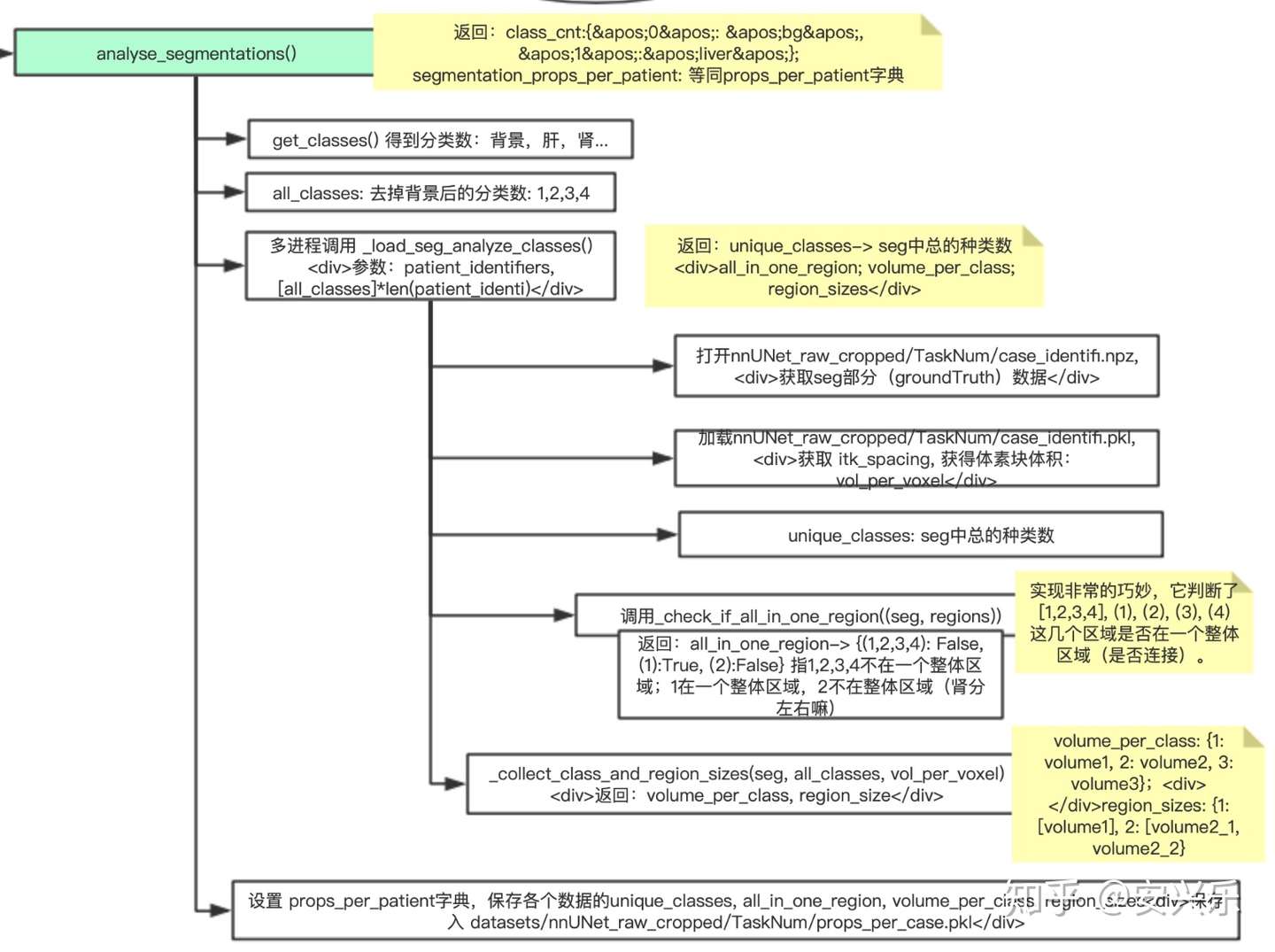
def analyse_segmentations(self):
class_dct = self.get_classes()
all_classes = np.array([int(i) for i in class_dct.keys()])
all_classes = all_classes[all_classes > 0] # remove background
if self.overwrite or not isfile(self.props_per_case_file):
p = Pool(self.num_processes)
res = p.map(self._load_seg_analyze_classes, zip(self.patient_identifiers,
[all_classes] * len(self.patient_identifiers)))
p.close()
p.join()
props_per_patient = OrderedDict()
for p, (unique_classes, all_in_one_region, voxels_per_class, region_volume_per_class) in \
zip(self.patient_identifiers, res):
props = dict()
props['has_classes'] = unique_classes
props['only_one_region'] = all_in_one_region
props['volume_per_class'] = voxels_per_class
props['region_volume_per_class'] = region_volume_per_class
props_per_patient[p] = props
save_pickle(props_per_patient, self.props_per_case_file)
else:
props_per_patient = load_pickle(self.props_per_case_file)
return class_dct, props_per_patient
get_classes得到分类字典class_dct:{0:’backgroud’, 1:’liver’…},分类数all_classes:[1, 2,3]。之后多进程调动_load_seg_analyze_classes(),这也是该类最为重要的函数!主要对体素、分割种类、数据分布进行统计。代码并不复杂:
def _load_seg_analyze_classes(self, args):
patient_identifier, all_classes = args
seg = np.load(join(self.folder_with_cropped_data, patient_identifier) + ".npz")['data'][-1]
pkl = load_pickle(join(self.folder_with_cropped_data, patient_identifier) + ".pkl")
vol_per_voxel = np.prod(pkl['itk_spacing'])
unique_classes = np.unique(seg)
# 4) check if all in one region
regions = list()
regions.append(list(all_classes))
for c in all_classes:
regions.append((c, ))
all_in_one_region = self._check_if_all_in_one_region((seg, regions))
# 2 & 3) region sizes
volume_per_class, region_sizes = self._collect_class_and_region_sizes((seg, all_classes, vol_per_voxel))
return unique_classes, all_in_one_region, volume_per_class, region_size
这里打开的nnUNet_raw_cropped目录下的case_xxx.*npz文件,获取mask(groundTruth)数据,得到unique_classes数,以及all_in_one_region字典:{((1,):True,(2,):False)…},2为肾脏,自然不在一个区域了;还有vol_per_voxel(每个体素的体积)。这样最后返回unique_classes, all_in_one_region, volume_per_class, region_size这四个变量。
接下来的回到analyse_segmentations()部分,将四个变量写入props_per_patient字典中,并save_pickle写入nnUNet_raw_cropped的props_per_case.pkl文件中。
“继续出栈”,过了好久我们再次回到了analyze_dataset()函数中。剩下的就是统计分布的代码了,统计size_reductions、modalities,然后直接写入dataset_properties字典中,调用save_pickle将数据属性保存入nnUNet_raw_cropped/TaskNUM/dataset_properties.pkl中。
原流程图文件请登录processon.com参阅。DatasetAnalyzer类流程图
